内容
Getting StartedとFilters and Editorチュートリアルを理解していれば、このチュートリアルによりCytoscapeを用いた発現解析の基礎を身につけられます。このチュートリアルでは以下を示します:
- 発現解析の入力フォーマット
- 発現データの値によるノードの色分け
- 生物学的なネットワークに基づいた発現データの評価
このチュートリアルと付随する講義はCSC, the Finnish IT center for scienceから提供されています。背景となる講義スライドと付随する映像資料は、CSC 提供の http://www.csc.fi/english/research/sciences/biosci... が利用可能です。
このチュートリアルでは以下のデータファイルを取り上げています:
- galFiltered.sif Cytoscape testData ディレクトリにあります。このネットワークには酵母のガラクトース代謝についてのタンパク質−タンパク質相互作用とタンパク質−DNA相互作用があります。
- galExpData.pvals Cytoscape testData ディレクトリにあります。このファイルには3つのpertubation experimentsの遺伝子発現値があります。個々の実験において、キーとなるタンパク質のレベルは人工的に摂動されています。
- galExpData.mrna Cytoscape testData ディレクトリにはありません。このファイルには galExpData.pvals データのサブセットが含まれています。
これらのデータセットの詳細は Science 2001, 292:929-34. を参照のこと。解析を始める前に、リンクを右クリックしてローカルディスクにデータをダウンロードしてください。ファイル名の拡張子が ".txt" になってないことを確認してください。
ここ: WEB START (ダウンロードサイズ約: 22 MB) をクリックして解析を始めてください。これにより我々のウェブサイトからプログラムとアノテーションデータをダウンロードし、あなたのコンピューター上でCytoscapeが開始します。
ここで、発現データをCytoscapeのネットワークに適用することにより、基本的な構造の探索を始めましょう。この節では、Cytoscapeにおける発現データのデータフォーマットを解説します。発現値によるネットワークのノードを規定するためものです。
ファイルの構造は以下の通りです: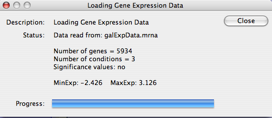
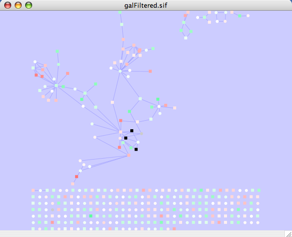
- 1. Cytoscapeを開始し、galFiltered.sif ネットワークを読み込みます。Data PanelとResults Panelを切り離したあとに、canvasを最大化し、. After detaching Data Panel and Results Panel, maximizing the canvas, and applying the spring-embedded layout ,下のようなネットワークが見られます。

- 2. お手持ちのテキストエディタを使って galExpData.mrna ファイルを開いてください。最初の数行は以下の通りです:
GENE COMMON gal1RG gal1RG gal80R
YHR051W COX6 -0.034 -0.034 -0.304
YHR124W NDT80 -0.090 -0.090 -0.348
YKL181W PRS1 -0.167 -0.167 0.112
YGR072W UPF3 0.245 0.245 0.787
ファイルの構造は以下の通りです:
- 最初の行はラベルです。
- すべての列は一つの空白文字かTABで区切られています。
- 最初の列はノード名であり、ネットワークのノード名と一致している必要があります!
- 第二列は遺伝子座名です。この列はオプションであり、Cytoscapeでは現在用いられていませんが、多くのマイクロアレイ解析パッケージの出力形式に則っています。ファイルの内容を読みやすくするためにあります。
- 残りの列は実験値です。一つの列が一つの実験であり、一行が一つのノードです。この例の場合、一つのノードに三つの発現値があります。
- 3. Fileメニューから Import → Attribute/Expression Matrix... を選択し、galExpData.mrna ファイルを読み込んでください。簡単な読み込みの後、ステータスウィンドウが開き、実験条件がいくつあるか(この例では三つ)、どの種類の有意な値が含まれているか(この例ではなし)が示されます。閉じるボタンをクリックしてください。

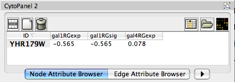
- 4. ここで、発現データをを見るために Node Attribute Browser を以下のように用います。
- Cytoscape canvas 上でノードを選択します。
- Node Attribute Browserで Select Attributes ボタンをクリックし、左クリックにより、gal1RGexp, gal4RGexp, and gal80Rexp の属性を選択してください。右クリックでメニューを閉じます。
- Node Attribute Browser にて、発現値のリストとしてノードを見ることができます。

Cytoscapeで発現データを利用する場合たいていは、 ネットワーク内のノードの visual attribute を発現値にセッティングすることでしょう。これにより強力なビジュアル化が可能となり、機能の関係や実験値の反応を同時に表現することができます。ここで、以下の手順を踏んでください。
- 1. このアイコンをクリックして VizMapper を開いてください:

- 2. Copy existing Visual style ボタンをクリックしてデフォルトのスタイルをコピーすることにより、 Gal80 という名前の新しい visual スタイルを作成してください。
- 3. 以下のように visual スタイルのノード色を定義してください:
- i. Node Color タブにより、Mapping Type を続けて設定してください。
- ii. ノード色のドロップダウンリストにて、属性 gal80RGexp を選択してください。Gal80の発現値による連続的な色をノードに割り当てます:
- (高い抑制を示す)大きな負の値は赤
- (低い抑制を示す)小さな負の値はピンク
- 0に近い値は白
- (低いinductionを示す)小さな正の値はライトグリーン
- (高いinductionを示す)大きな正の値はbright green
- 外れ値(<-2.5の負の値と>2.1の正の値)はそれぞれ青と黒
- iii. デフォルトのノード色のピンクがこの連続色の中に含まれていることに気をつけてください。発現値のないデータと低い抑制を示す値を区別するために、この連続色以外の色を選択することがよく行われます。Change Defaultをクリックしてデフォルト色として灰色を選択します。
- 4. Click on Apply. 多くのピンク、緑、白のノードといくつかの灰色のノードそして黒いノードを目にするでしょう。
このセクションでは発現データがネットワークデータとどう結合され生物学的に何を言えるのか、のシナリオの一つを示します。
最初に、あなたのデータの背景を示します。あなたは酵母の研究をしており、酵母の転写因子であるGal1、Gal4、Gal80遺伝子の研究をしているとします。あなたの発現の実験はすべて、これらの転写因子遺伝子の some pertubation を含むとします。Gal1、Gal4、Gal80は相互作用ネットワーク内で表され、酵母のローカスタグとしてそれぞれGal1はYBR020W、Gal4はYPL248C、Gal80はYML051Wとラベルされています。
ネットワークにはタンパク質ータンパク質(pp)相互作用とタンパク質ーDNA(pd)相互作用が含まれます。ここで、タンパク質ータンパク質相互作用を除外しタンパク質ーDNA相互作用に着目することとします。

すべての3つの黒いノード(highly induced)はグラフの同じ領域にあることに注意してください。詳細を見るためにはグラフを拡大してください。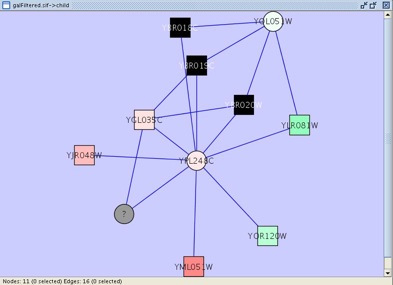
やったぜ!ネットワーク解析と発現データは力強い組み合わせであり、ここでかなりの解析力を身につけましたね。おいしいコーヒーにでもどうぞありついてください。
コメントや提案があれば cytoscape-discuss メーリングリストに投稿してください。
Return to Cytoscape introductory tutorials.
最初に、あなたのデータの背景を示します。あなたは酵母の研究をしており、酵母の転写因子であるGal1、Gal4、Gal80遺伝子の研究をしているとします。あなたの発現の実験はすべて、これらの転写因子遺伝子の some pertubation を含むとします。Gal1、Gal4、Gal80は相互作用ネットワーク内で表され、酵母のローカスタグとしてそれぞれGal1はYBR020W、Gal4はYPL248C、Gal80はYML051Wとラベルされています。
ネットワークにはタンパク質ータンパク質(pp)相互作用とタンパク質ーDNA(pd)相互作用が含まれます。ここで、タンパク質ータンパク質相互作用を除外しタンパク質ーDNA相互作用に着目することとします。
- 1.ppにマッチするテキスト属性に関するエッジを選択するフィルターを作成します。詳細はfilters and editingチュートリアルを参照してください。
- 2.フィルターを適用します。362個のエッジのうち21個を選択します。
- 3.Editメニューにて、Delete Selected Nodes and Edgesを選択し、残ったエッジを見るためにグラフレイアウトを適用します。yFiles Organicレイアウトを適用するとネットワークは下図のようになります:

すべての3つの黒いノード(highly induced)はグラフの同じ領域にあることに注意してください。詳細を見るためにはグラフを拡大してください。
- 4.3つの黒いノードのうち、YPL248CとYOL051Wの2つのノードに注目してください。この2つのノードと近隣のノードを選択し、新しいネットワークにコピーしてください。これにより、そのノードを含む相互関係がわかりやすくなります。レイアウトやズームを工夫すると、この新しいネットワークは下記のようになります:

- 5.node attribute browserでの探索ののち、以下のことがわかるでしょう:
- すべての黒い3つのノードに関係する2つのノードはYOL051W (Gal11, a general transcription cofactor with many interactions)とYPL248C (Gal4)である。
- 両方のノードは発現の変化は小さく、統計的に優位なほどは変化していない。これは薄い色の円として表示されている。 These slight changes in expression suggest that the critical change affecting the black nodes might be somewhere else in the network, and not either of these nodes.
- YPL248CはYML051W (Gal80)と関係し、有意なレベルの抑制を示している:これは赤い四角で示されている。
- 一方、誘導を高いレベルで示すYPL248C遺伝子と関係するほとんどのノードYML051Wは有意な抑制の証拠を示していることに注意すること:これらは緑か黒の四角で示されている。
- 6.NCBIのウェブサイト( http://www.ncbi.nlm.nih.gov/ )に行き、Gene databaseでYPL248Cを検索してください。Gal4を含む結果が得られるでしょう。Gal4のリンクをクリックして詳細な情報を得てください。
- 7.Gal4の説明を読むと、それがGal80によって抑制を受ける転写因子であることがわかるでしょう。
- 8.Gal4の転写活性化はGal80によって抑制を受けることが、ここでの解析でわかります。そう、Gal80の抑制はGal4の転写活性化を促進します。Gal4の発現を通してGal4自身はそれほど変化を見せず、Gal4転写物はGal80が抑制されるときの活性転写因子と見なされる。これは、Gal4の近辺で強いアップレギュレーションを示すことの説明となっている。
やったぜ!ネットワーク解析と発現データは力強い組み合わせであり、ここでかなりの解析力を身につけましたね。おいしいコーヒーにでもどうぞありついてください。
コメントや提案があれば cytoscape-discuss メーリングリストに投稿してください。
Return to Cytoscape introductory tutorials.