スライドショー Filtering and Editing in Cytoscape (15 分)
ハンドアウト Filtering_and_Editing_in_Cytoscape.pdf (5 ページ)
チュートリアルのソース Cytoscape Wiki
チュートリアルのキュレーター Kristina Hanspers
このチュートリアルではCytoscapeのいくつかの応用を紹介します:
ハンドアウト Filtering_and_Editing_in_Cytoscape.pdf (5 ページ)
チュートリアルのソース Cytoscape Wiki
チュートリアルのキュレーター Kristina Hanspers
目次
1 前提条件
2 ネットワークと属性の読み込み
3 フィルターの利用
4 編集
このチュートリアルではCytoscapeのいくつかの応用を紹介します:
- フィルタを適用して信頼性の低いエッジを除去します。
- Cytoscapeのグラフエディタを用いて基本的な編集を行います。
このチュートリアルでは以下のプラグインとデータセットを扱います:
- Institute for Systems BiologyのRowan Christmas作成のNetwork Filterプラグイン。
- RUAL.subset.sif、2005 Oct 20 in Rual et al., Nature 437(7062):1173-8にヒト相互作用データセットとして発表されたものの一部でありCytoscapeから利用可能です。
- 属性セットRUAL.naも同じサイトから利用可能です。
- Cytoscapeを開始します。Cytoscapeのダウンロードとインストールの詳細はGetting Started(訳注:古いチュートリアルへのリンクとなっています)を参照のこと。
- CytoscapeのFileメニューからImport → Network (Multiple file types)...を選択し自分でダウンロードしたファイルパスを指定してRUAL.subset.sifネットワークファイルを読み込みます。
- File → Import → Node Attributes...からノードの属性RUAL.naファイルを読み込みます。
- yFiles organic レイアウトをネットワークに適用します。
ノードの属性は以下の様になります:
- ノード属性ファイルでオフィシャルノード名として読み込んだものはオフィシャルと呼びます。この属性をどう指定されているか、RUAL.naファイルを普段使うテキストエディタで開いてみてください。
- Cytoscapeデスクトップでは、Node Attribute Browserにて、Select Attributes
 ボタンをクリックしてみてください。
ボタンをクリックしてみてください。 - Official HUGO Symbolと名付けられた属性に注目してください。マウスの左ボタンでクリックしてください。
- マウスの右ボタンでメニューが現れます。
- Node Attribute Browserに2つの列があります、1つはラベルがIDとなっていて一方はOfficial HUGO Symbolとなっています。Cytoscape canvasでいくつかのノードを選択してみてください。選択したノードのID(この例ではEntrez gene ID)とオフィシャルな遺伝子名が表示されます。
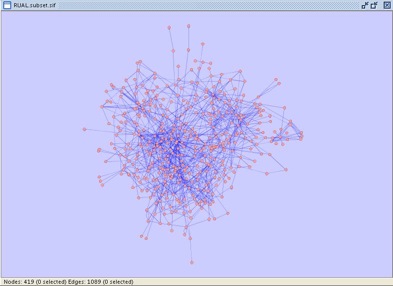
あなたの扱うネットワークにはいろいろなタイプのエッジが含まれています:Y2H yeast two-hybrid interactions、coAP GST pull-down interactions、three types of literature-based interactions、信頼性が増えるにつれ:non-core、core、hypercoreとなります。Edge Attribute Browserでエッジタイプを試すことにより確認することができます。エッジを選択することを思い出してください、CytoscapeデスクトップのSelectメニューをまず最初に開きMouse Drag Selects...を選択します。ここでCytoscapeのフィルターを用いて低い信頼性のnon-coreエッジを除去します。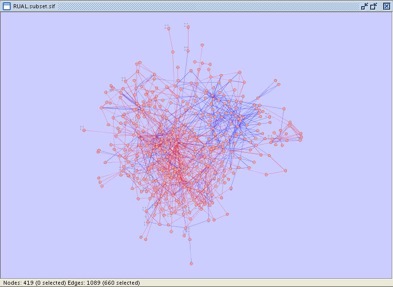
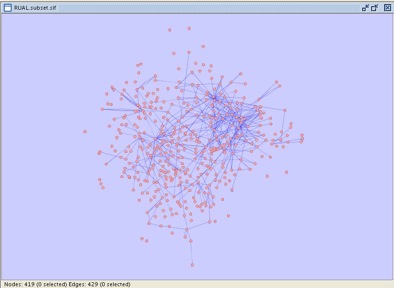
最初のネットワークと比べるとエッジ数が減っていますが、実験的に確認されたものか文献に基づいて高い信頼性のあるものは残っています。解析の手法により、より適切なエッジのセットとなります。エッジのないノードが複数残りますが、フィルターで除去したくなるでしょう。ここではそのひとつの方法を示します: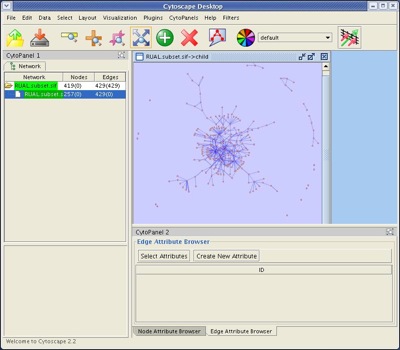
セルフノードのみを持つノードは除去できないことに注意してください。しかしこの点は、マウスでクリックして簡単に削除できます。
- CytoscapeのデスクトップメニューからSelect → Use Filtersを選択します。Control Panel内にFiltersウィンドウが開きます。
- Filtersウィンドウ内で、OptionsドロップダウンをクリックしCreate new filterを選択します。(訳注:New fileter nameポップアップに新しいフィルターの名前を入力します。)
- Filter Definitionフィールドで、ドロップダウンからedge.interactionを選択します。Addボタンをクリックします。
- Advancedフィールド内のinteractionフィールドにてドロップダウンからnon_coreを選択します。Applyをクリックしてフィルターを適用します。(訳注:ApplyではなくApply Filter)
- Use Filtersウィンドウを閉じ(約注:とくに閉じるべきウィンドウはない)、660エッジが選択されていることを確認します(訳注:Filtersの一番下のEdgesの()内の数が660となっています)。Cytoscapeウィンドウは下記のようになっているでしょう。

- CytoscapeデスクトップのEditにて、Delete Selected Nodes/Edgesを選択します。
- Cytoscapeウィンドウは下記のようになっているでしょう:

最初のネットワークと比べるとエッジ数が減っていますが、実験的に確認されたものか文献に基づいて高い信頼性のあるものは残っています。解析の手法により、より適切なエッジのセットとなります。エッジのないノードが複数残りますが、フィルターで除去したくなるでしょう。ここではそのひとつの方法を示します:
- テキストの属性相互作用に関するエッジのタイプを選択するフィルターを作成し、ワイルドカードの * を指定します。先程のフィルターと同様の方法で作成できまが今回はAdvancedフィールド内のinteractionフィールドのnon_coreの代わりに * と入力します。このフィルターはcanvas上のすべてのエッジを選択します。Apply(訳注:Apply Filter)をクリックし、すべてのエッジが選択されることを確認してください。
- FileからNew → Network → From selected nodes, selected edgesを選択します。これにより257ノード、429エッジの"child"ネットワークが作成されます。
- spring-embeddedレイアウトを適用します(訳注:Layout-Cytoscape Layouts-Spring Embedded)。これにより以下のネットワークが作成されます。

セルフノードのみを持つノードは除去できないことに注意してください。しかしこの点は、マウスでクリックして簡単に削除できます。
時には、ネットワークをすこし修正するほうが役立つことがあります:ノードやエッジを追加したり、削除したり。例えば、生物学的プロセスの知見を最優先と考えている場合、そのプロセスで既知のタンパク質があり、自分のデータセットにはない場合ノードを追加したくなるでしょう。この説では以下にその方法を示します。
- Cytoscapeデスクトップの左側のControl PanelにてEditorタブをクリックします。
- Add a Nodeと名付けられたノード上でマウスの左ボタンをクリックし、マウスの左ボタンを押したままcanvas上にドラッグすることによりcanvasにノードを追加します。
- マウスでノードを選択します。Node Attribute BrowserにてIDがnode0となっています、以下に示します:

- Give your new node a name by going to the Node Attribute Browserにて新しいノードに名前を付けます、そのノードのエントリーをクリックし、Official HUGO Symbolと名付けられた列で新しい名前を入力してください。ノードに新しいIDは入力できないことに注意してください。(訳注:EditorタブにてSpecify Identifier:にチェックを入れるとIDが入力可能となります)
- あたしいノードと既存のノードにエッジを追加するには以下のようにしてください:(訳注:Directed Edgeはなく、Ctrlキーを押しながらノードをクリックすると黒い線が現れ他のノードをクリックするとエッジとなります。Escキーで黒い線がキャンセルされます)
- Cytoscape EditorにてDirected Edgeをクリックします。
- Directed Edgeからマウスボタンを押したままcanvas上の新しいノードへドラックします。左ボタンを離すと2つのものが現われます:マウスがノード上にあるときは太い黒い縁が;ノード上ではない場合は太い黒い線となります。他のノードをクリックすると二つのノードの間に新しいエッジが現れます。
- 新しいノードと既存のノードの間にいくつかエッジを作ってみてください。
- 新しいノード(とそれに付いているエッジ)を削除するにはノードをマウスで選択し、(訳注:Editメニューから)Delete Selected Nodes/Edgesを選択します。ノードを削除した場合、その周りのエッジも削除されることを確認してください。