スライドショー Introduction to Cytoscape (30分)
ハンドアウト Introduction_to_Cytoscape.pdf (9ページ)
チュートリアルのソース Cytoscape Tutorial (Yeyejide Adeleye)
チュートリアルのキュレーター Anna Kuchinsky, Scooter Morris, Alex Pico
Cytoscapeはネットーワークについて情報付加、可視化、データ解析を行えるオープンソースのソフトウエアツールです。このチュートリアルはCytoscapeへのイントロダクションのパート2であり、以下をカバーします:
ハンドアウト Introduction_to_Cytoscape.pdf (9ページ)
チュートリアルのソース Cytoscape Tutorial (Yeyejide Adeleye)
チュートリアルのキュレーター Anna Kuchinsky, Scooter Morris, Alex Pico
目次
1 プラグインマネージャ
2 ネットワークとパスウェイのリソース
2.1 WikiPathways
2.2 Agilent Literature Search
2.3 非デフォルトデータのマッピング
2.4 ネットワークの拡張
3 その他のプラグインのデモ
3.1 プラグイン1:Vista Clara
3.2 プラグイン2:Cluster Maker
3.3 プラグイン3:BiNGO
Cytoscapeはネットーワークについて情報付加、可視化、データ解析を行えるオープンソースのソフトウエアツールです。このチュートリアルはCytoscapeへのイントロダクションのパート2であり、以下をカバーします:
- プラグインマネージャ
- ネットワークとパスウェイのリソース
- プラグインのデモ
プラグインマネージャとはユーザが早く容易に拡張機能をCytoscapeに追加するものであり、プラグインのインストールや更新のためにあちこちウェブサイトを探し回る必要をなくすものです。
もしインターネットに接続できないのならば、利用可能なプラグインのリストを見ることはできず、あるいは自動更新も不可ですが、すでにインストール済みのプラグインを見ることと削除は可能です。
新しいプラグインのインストール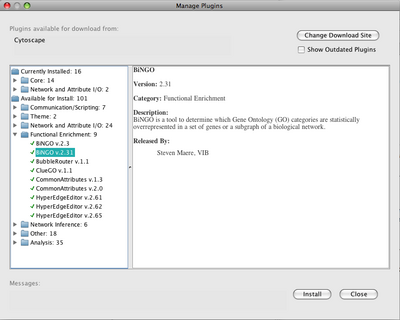
もしインターネットに接続できないのならば、利用可能なプラグインのリストを見ることはできず、あるいは自動更新も不可ですが、すでにインストール済みのプラグインを見ることと削除は可能です。
新しいプラグインのインストール
- Plugins -> Manage Pluginsを指定します。ポップアップウィンドウの左側のCurrently Installed and Available for Install''と書かれたプラグインフォルダーを見ましょう。サブフォルダーを見るにはダブルクリックをしてください。特定のプラグインの詳細を見るには、プラグイン名をクリックするとウィンドウの右側に基本的な情報が表示されます。

- Currently Installedフォルダーにはデフォルトのプラグインがあり、Cytoscapeの更新ごとに完全に更新され、同時に追加もされます
- Available for Installフォルダーにはインストール可能なプラグインが表示されます。
- jActiveModulesをインストールするには: Available for Installフォルダーを開き、Analysisフォルダーを選択し、jActiveModules v2.22(訳注:Cytoscape 2.81では最新はv2.23だがどのバージョンもCytoscape 2.8.1では動作しないとのこと)を選択します。ウィンドウの下のインストールボタンをクリックします
- Closeボタンをクリックして Plugin Manager を終了します
- Plugins menuを選択するとプラグインリストにjActiveModulesが現れます
- Cytoscapeのプラグインのページを開きます(http://cytoscape.org/plugins.html(訳注:原文では plugins となっていますがリンク切れ。正しくは plogins.html ))、
- 下にスクロールしてプラグインを見つけます
- ファイルダウンロードのリンクをクリックし、Cytoscape/plugins フォルダー内に保存します。
- Cytoscapeの再起動をするとプラグインがインストールされます。
しかしネットワークを持っていなければどうしたらよいでしょうか。この節ではCytoscapeとプラグインで用いるために、公共のリソースからネットワークとパスウェイを検索、構築、推測する方法を示します。
- Plugin ManagerからGPML-Pluginをダウンロードすることから始めましょう。Network and Attribute I/Oフォルダーにあります。
- File-> Import -> Network from web servicesを選択します
- ドロップダウンメニューからWikiPathways Web Service Clientを選択します
- 検索語に(‘TCA’)を指定し生物種として(‘Saccharomyces cerevisiae’)を選択します
- 関連のあるパスウェイのリストが結果として返ってきます
- ‘Glycolysis and Gluconeogenesis’をダブルクリックしてパスウェイをインポートします
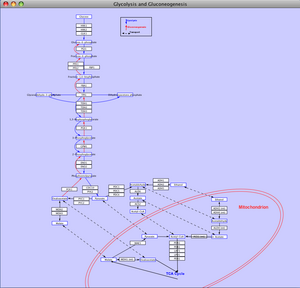
WikiPathways は GPML データフォーマットを用いており、注釈付された座標を持ち、グラフィカルなアノテーションとラベルがあり、加えて、ノードとエッジのネットワークの情報があります。アノテーションのレイヤーを隠すには View>Toggle GPML Annotations を選択し、Cytoscapeに用意された他のネットワークで処理します。 (訳注:何も変わらない気が、、、)
Agilent Literature Searchはテキストマイニング技術を用い、科学論文から取得した情報より"関連"ネットワークを生成するものです。これは遺伝子とタンパク質が病気や生物学的プロセスとどのように関連するかを理解するのに役立つものです。
- Agilent Literature Searchを開くにはPlugins->Agilent Literature Searchを選択します
- "Terms"に"pda1"と"adh1"を入力します
- "Context"に"glycolysis"と入力します
- Use Aliasesを選択します。シノニム"adh1"を確認しましょう
- Concept Lexiconで次を選択します: Saccharomyces cerevisiae
- Interaction Lexiconで次を選択します: "relaxed"
- 青いPlay buttonをクリックし、検索を実行します。Query Matches windowに検索結果が返ってきます。

インポート済みのネットワークとパスウェイにデータをマップするとき、キーとなるIDでデータとネットワークの両方を関連付けなければならない。このような場合Attribute Importerによりマッピングオプションを適用できる。
- File-> Import->Attribute from Table (Text/MS Excel)を選択します
- sampleDataフォルダからgalExpData.pvalsファイルを選択し、開くをクリックします。
- Advancedセクションで、Case Sensitiveのチェックを外します
- Advancedセクションで、Show Text File Import Optionsにチェックを入れます
- Delimiterセクションで、Spaceにチェックを入れます
- Attribute Namesセクションで、Transfer first line attribute namesにチェックを入れます
- Show Mapping Optionsにチェックを入れます
- “GENE”を“COMMON”にして、,“ID”を“canonicalName”に替えます
- Preview内の冗長な列名の2番目のヘッダーをクリックし、読み込みから除去するか右クリックで名前を変えます
- Importをクリックします
- ここでControl PanelのVizMapperタブを開き、Node Colorプロパティを選択します。ダブルクリックしてアクティブにします。
- “gal1RG”を選択し、“Continous”を選択します
- 色の階調のところをクリックし、Gradient Editorを開きます
- 端点の色を設定し、色の階調を作ります
- 読み込んだすべてのパスウェイとネットワーク上にデータが可視化されました!

このプラグインでも対話式の特徴を備えており、文献検索の結果を用いてネットワークを拡張することができます。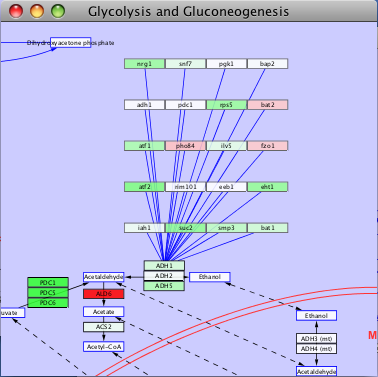
- 読み込んだ“Glycolysis and Gluconeogenesis”パスウェイを開き、検索ボックスで“ADH1”を検索します
- ノード上で右クリックしてEvidence from Literature > Extend Network from Literatureを選択します (訳注:私の環境ではこのメニューは表示されず)
- ADH1の近くのノードで新たに選択とドラックをして、Layout > CyLayouts > Grid > Selected Onlyを選択します。ADH1に近くの領域をグリッドレイアウトにします

- データマッピングが自動に行われることに注目してください
- 新しいノードのエッジ上で右クリックをしてShow Sentences from Literatureを選択します
- この方法により、文献検索とデータの可視化に基づいて既知のパスウェイを拡張することができます
もし上記のチュートリアルから続けているなら、ここでCytoscapeを再起動してネットワークとノードと属性をきれいな状態にしてください。以下のプラグインはすべて同じデモファイルを用います:
- File-> Openを選択します (現在のセッションを削除しますか と聞いてくるので「はい」をくりっくします)
- Open a Session File ダイアログが開きます
- sampleDataフォルダからgalFiltered.cysファイルを選択し「開く」をクリックします
使用方法:対話式にてデータを探し、表形式で表します
VistaClaraプラグインはPlugin Manager (1.3節参照)から利用できます(訳注:"Analysis"フォルダ内にあります)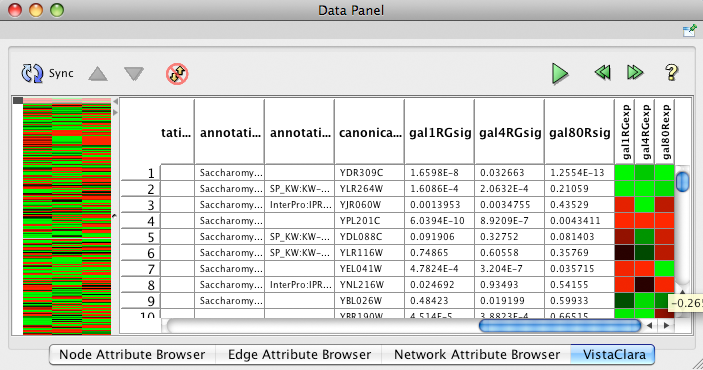
左側に自動的に表示される最初の列はデータのヒートマップです。最後の列に表示されるのは選択されたデータについてのヒートマップの値です。注意点:いくつかの列(例:gal80R)は認識されていないか、正しいフォーマットではありません。
VistaClaraプラグインはPlugin Manager (1.3節参照)から利用できます(訳注:"Analysis"フォルダ内にあります)
- 画面下部のData PanelにてVistaClaraタブを選択します
- syncをクリックします
- Data Panelを広げるかundockすると使いやすいでしょう

左側に自動的に表示される最初の列はデータのヒートマップです。最後の列に表示されるのは選択されたデータについてのヒートマップの値です。注意点:いくつかの列(例:gal80R)は認識されていないか、正しいフォーマットではありません。
- データのいくつかの列のフォーマットを修正するために列のヘッダーを右クリックし、正しいフォーマットを選択します。‘log2 ratio’をgalFilteredデータセットのすべての fold 値のフォーマットとして試してみてください。
- ソートするには列の上で右クリックして sort を選択するとソートされます
- ヒートマップ上で選択かドラックをすると値がスクロールします
- ヒートマップデータのカラムを選択するとネットワーク上にて値を色分けで表示します
- Click the playボタンをクリックするとすべてのヒートマップ列の値を周期的にネットワークに表示します
- VizMapperにて‘VistaClara’visual style が作成されていることに注目してください。以前の visual styles を選択すればいつでも元のスタイルに戻せます。
clusterMakerプラグインはCytoscapeのノードとエッジ属性についてクラスタリングやビジュアルなクラスター作成を行う共通のフレームワークを提供します。clusterMakerの利用法の重要なものの一つとしては階層的クラスタリングあるいはk-meansクラスタリングを用いた発現データの解析をサポートすることがあげられます。他の例ではepistatic mapping (E-MAP) データの解析を含み、同様にタンパク質ファミリーネットワークのクラスタリングにより、予測したタンパク質ファミリーにノードを割り当てることなどがあります。
使用法:クラスター解析の実施と可視化
ClusterMakerプラグインはPlugin Manager (1.3節参照)から利用できます (訳注:Analysisフォルダ内にあります)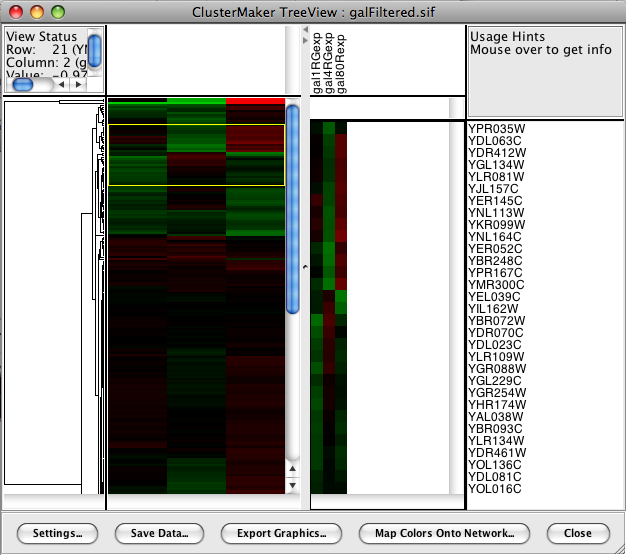
これは標準的なクラスタービューであり完全なヒートマップが左にあります。選択したヒートマップは中間に表示され遺伝子名は右側に表示されます。
使用法:クラスター解析の実施と可視化
ClusterMakerプラグインはPlugin Manager (1.3節参照)から利用できます (訳注:Analysisフォルダ内にあります)
- PluginsメニューからCluster->Hierarchical clusterを選択します
- ctrlキー (Macでは cmd キー) を使って3つのfold値データ (gal1RGexp, gal4RGexp, gal80Rexp) を選択します
- Only use selected nodes/edges for clusterをクリックしてチェックを外します (訳注:デフォルトでチェックが外れています)
- Create Clustersをクリックします
- Visualize Clustersをクリックします

これは標準的なクラスタービューであり完全なヒートマップが左にあります。選択したヒートマップは中間に表示され遺伝子名は右側に表示されます。
- Settings…をクリックし、コントラスト、色の階調、データ範囲を調整します
- クリックとドラックでヒートマップ上の行を選択します;Shiftキーとクリックでヒートマップ上の列毎に選択します
- 列を選択しMap Colors Onto Network…をクリックするとクラスターのヒートマップからヴィジュアルスタイルを作成し、ネットワーク上のデータのヴィジュアル化を行います
- ヒートマップ上で列を選択してMap Colors Onto Network…をクリックした場合、見たい列を選択するポップアップダイアログが現れます;3つすべてを選択してAnimate Vizmapをクリックします
- ここでズームアウトしてネットワークを見るとデータが生きているように見えるでしょう!このビューによりネットワーク上の“hot spots”が見つけられるでしょう。アニメーションを止めるにはStop Animationをクリックします。そしてVizMapperを使って元のビジュアルスタイルに戻してください。'''
使用法:過剰発現解析の実行と可視化を行います
BiNGOプラグインはPlugin Manager (1.3節参照)から利用できます (訳注:Functional Enrichmentフォルダ内にあります)
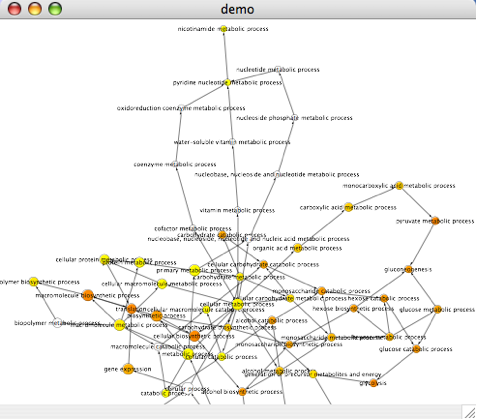
BiNGOプラグインはPlugin Manager (1.3節参照)から利用できます (訳注:Functional Enrichmentフォルダ内にあります)
- プラグインメニューからBiNGOを選択します
- ネットワークからノードのサブセットを選択します、例えば
- ClusterMaker解析から得られたクラスターを選択する
- Data Panelでソートかフィルターを掛けられたデータ値に基づいて選択する
- クラスターに名前をつける
- 他の設定を確認します (このチュートリアルでは任意です)
- ‘Start BiNGO’をクリックします
- ランク付けされたGO termと統計値の表と有意性により色付けされたGO termのネットワーク、色の階調の凡例のウィンドウが表示されます

- クラスターの選択とBiNGOパラメーターを扱ってあなたのデータの解析の可能性を探検してください。