�ǽ�������
![]() bioinformatics 2011ǯ05��31��(��) 18:05:39����
bioinformatics 2011ǯ05��31��(��) 18:05:39����
FrontPage-Cytoscape-Tutorials
������Node Attribute Browser��Ȥ��ʲ��Τ褦��ȯ���ǡ�����������ޤ���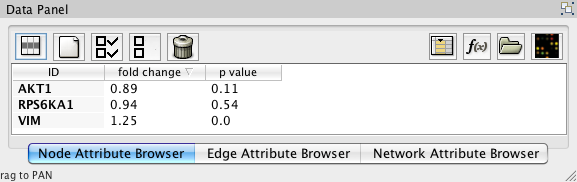
���饤�ɥ��硼 Basic Expression Analysis in Cytoscape (30 ʬ)
�ϥ�ɥ����� Basic_Expression_Analysis_in_Cytoscape-Human.pdf (11 �ڡ���)
���塼�ȥꥢ��Υ���졼���� Kristina Hanspers, Alex Pico, Mike Smoot
3.3 �Ρ��ɤη������� �� 3.4 �Ρ��ɤζ��������꤬���ԤδĶ��Ǥ�ȿ�Ǥ��줺������ߥ��β�ǽ�����⤯���ȤǸ�ľ���ޤ���
Cytoscape�ϥ����ץ������եȥ������ʥץ�åȥե�����Ǥ��ꡢ�ͥåȥ���������ղá��Ļ벽��������Ԥ���ΤǤ������Υ��塼�ȥꥢ��Ǥϰʲ���Ҳ𤷤ޤ���
�ϥ�ɥ����� Basic_Expression_Analysis_in_Cytoscape-Human.pdf (11 �ڡ���)
���塼�ȥꥢ��Υ���졼���� Kristina Hanspers, Alex Pico, Mike Smoot
�ܼ�
1 �ͥåȥ�����ɤ߹���
2 ȯ���ǡ������ɤ߹���
3 ȯ���ǡ����βĻ벽
3.1 �Ρ��ɿ�������
3.2 �ǥե���ȤΥΡ��ɿ�������
3.3 �Ρ��ɤη�������
3.4 �Ρ��ɤζ���������
4 �ǡ������ϵ�ǽ
4.1 �Ρ��ɤΥե��륿��
4.2 �Ρ��ɤθ���
4.3 �Ρ��ɤ�õ��
3.3 �Ρ��ɤη������� �� 3.4 �Ρ��ɤζ��������꤬���ԤδĶ��Ǥ�ȿ�Ǥ��줺������ߥ��β�ǽ�����⤯���ȤǸ�ľ���ޤ���
Cytoscape�ϥ����ץ������եȥ������ʥץ�åȥե�����Ǥ��ꡢ�ͥåȥ���������ղá��Ļ벽��������Ԥ���ΤǤ������Υ��塼�ȥꥢ��Ǥϰʲ���Ҳ𤷤ޤ���
- �ۤʤ륽��������Υǡ����η�硧�ޥ��������쥤ȯ���ǡ�������μ¸��ͤ���ߺ��ѥǡ��������ǤΥͥåȥ���ǡ�����
- ȯ���ǡ������Ѥ����ͥåȥ���βĻ벽��
- ȯ���ǡ����˴�Ť��ͥåȥ���Υե��륿���
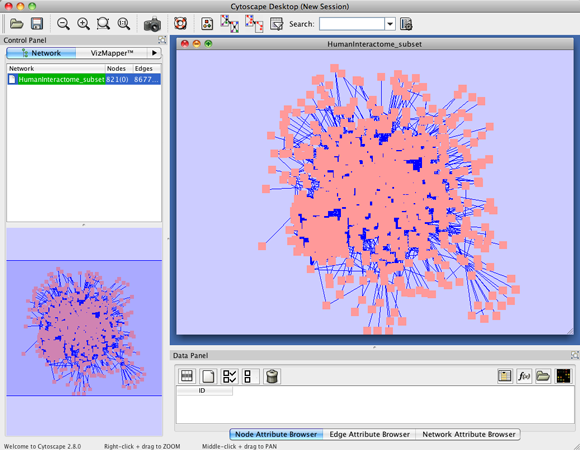
- Cytoscape�Ϥ�File:HumanInteractome subset.sif�ͥåȥ�����ɤ߹��ߤޤ���
- force-directed�쥤�����Ȥ�Ŭ�Ѥ��Ρ��ɤΥ쥤�����Ȥ��ȿ������ޤ���"Layout->Cytoscape Layouts->Force-Directed Layout"��˥塼�����ޤ���
- �ͥåȥ���Ϥ��Τ褦�ˤʤ�ޤ���

- ȯ���ǡ���File:Pellegrini et al Data.txt.zip�����������ɤ�unzipŸ���ޤ���
- ������Υƥ����ȥ��ǥ�����Pellegrini_et_al_Data.txt�ե�������ƤߤƤ����������ǽ�ο��Ԥϰʲ��ΤȤ���ȤʤäƤ��ޤ���
Gene Symbol Entrez id Probeset CREB kd control p value fold change Sign CREB binding���Υե�����ˤĤ��ưʲ��ξ�����ǧ���Ƥ���������
A2M 2 217757_at 42.24 57.45 0.4 1.5 yes
A2ML1 144568 1553505_at 67.95 206.47 0.11 0.58
A2ML1 144568 1564307_a_at 160.05 183.44 0.81 0.95
- �ǽ�ιԤϥ�٥뤫��ʤ�ޤ���
- ���٤Ƥ����1�Ĥ�TAB�Ƕ��ڤ��Ƥ��ޤ���
- �ǽ����ϥΡ���̾�Ǥ��ꡢ�ͥåȥ���ΥΡ���̾�����Τ˰��פ���ɬ�פ�����ޤ���
- 2���ܤ�Entrez Gene ID�Ǥ���
- 3��̾��Affymetrix probe set ID�Ǥ���������ϥ��ץ����Ǥ��ꡢ���ߤ�Cytoscape�ǤϻȤ��Ƥ��ޤ���¾�Υޥ��������쥤���ϥġ���Ǥβ��ϤǤ�ͭ�ѤǤ���
- �Τ������ϼ¸��ͤǤ����¸���ʿ��ȯ���͡�����ȥ����뽸�Ĥ�ʿ��ȯ���͡���Ӥ�p�ͤ���Ψ�Ѳ���CREB�ǡ����ӥ�ˤ����ʤ��˴ؤ�餺�����ʤäƤ��ޤ����ǡ��������ξܺ٤ˤĤ��Ƥ�manuscript�ȤΤ��ȡ�
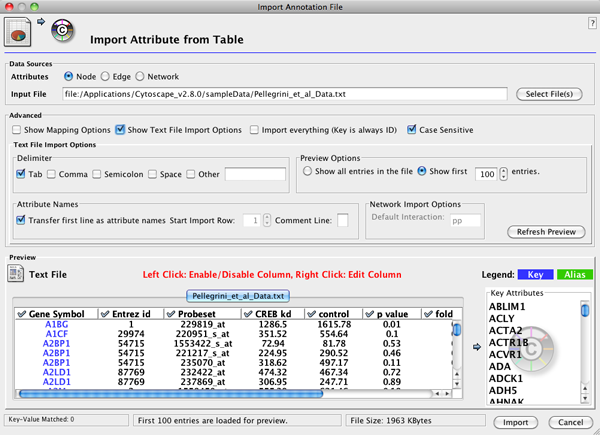
- File��˥塼���顢Import �� Attribute from Table (Text/MS Excel)�����ޤ���
- °���Υ����פ�"Node"��å����������ǥե�����͡ˤ��ޤ���
- Pellegrini_et_al_Data.txt�ե���������ޤ���
- "Text File Import Options"��������Show Text File Import Options�˥����å��ܥå�����å����ޤ���
- "Delimiter"����������"Tab"�����å��ܥå����������å�����Ƥ��뤳�Ȥȡ�"Delimiter"���¾�Υ����å��ܥå����������å�����Ƥ��ʤ����Ȥ��ǧ���ޤ����ץ�ӥ塼�˥ǡ�����ʣ������ɽ������Ƥ��ޤ���
- "Attribute Names"����������"Transfer first line as attribute names"�����å��ܥå�����å����ޤ����ץ�ӥ塼�ˤƺǽ�ιԤ���̾�ȤʤäƤ��ޤ���
- "Import"�ܥ����å�����°���ǡ������ɤ߹��ߤޤ���

������Node Attribute Browser��Ȥ��ʲ��Τ褦��ȯ���ǡ�����������ޤ���
- Cytoscape canvas��ǥΡ��ɤ�å��������ޤ���
- Node Attribute Browser�ˤ�Select Attributes�ܥ���
 ��å�����������å��ˤ�ä�°��"fold change"��"p value"�����ޤ���������å��ǥ�˥塼���Ĥ��ޤ���
��å�����������å��ˤ�ä�°��"fold change"��"p value"�����ޤ���������å��ǥ�˥塼���Ĥ��ޤ��� - Node Attribute Browser�ˤ�ȯ���ͤ˴ؤ��ƥΡ��ɤ��ꥹ�Ȥ��졢���Τ褦�ˤʤ�ޤ���

Cytoscape�Ǥ�ȯ���ǡ����ΰ���Ū�ʻȤ�����ϡ�ȯ���ǡ����˴ؤ���ͥåȥ����ǤΥΡ��ɤΥӥ��奢���°�������ꤹ�뤳�ȤǤ��礦������ˤ�궯�ϤʲĻ벽����ǽŪ��Ϣ��ɽ�����¸�Ū��ȿ����Ʊ����ɽ�����Ȥ��Ǥ��ޤ��������ǡ����������ɤäƸ��Ƥ����ޤ��礦��
- Visual Mapping Browser���Unused Visual Properties���������ˤ���Node Color����֥륯��å����ޤ���
- ����ˤ��Node Color��Visual Mapping Browser�Υȥåפ˰�ư���ޤ���
- Node Color����������"Please select a value!"��å����ޤ���
- ���ѽ����°��̾���ɥ��åץ������˥塼�Ǽ�����ޤ���"fold change"�����ޤ���
- Node Color����������"Please select a mapping"��å����ޤ���
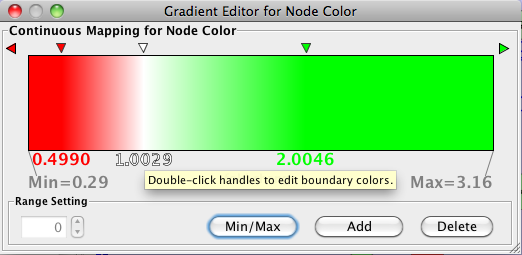
- ���Ѳ�ǽ�ʥޥåԥ����פ��ɥ��åץ������˥塼�Ǽ�����ޤ���"Continuous Mapping"�����ޤ���
- ����ˤ���������ؤδ���Ū�ʿ��γ�Ĵ��������ޤ���
- �����Ѥ��뤿��˿��γ�Ĵ��å����ޤ�������ˤ�곬Ĵ�Խ��Υ����������ݥåץ��åפ�ɽ������ޤ���
- �㤤�����ͤο����Ѥ��뤿��˺�ü�ι������Ѥ���֥륯��å����ޤ������뤤�֤����ޤ���
- ³����2���ܤι������Ѥ�0.5�˶ᤤ�ͤޤǥ��饤�ɤ����ޤ�������ˤ���֤�����ޤǤγ�Ĵ�ˤʤ�ޤ�������������Τ褦���֤�����ˤ���ˤϡ����λ��Ѥ���֥륯��å��������뤤�֤�����ɬ�פ����
- ��ü����������ü����2���ܤΤϤ��ˤ����Ѥ饤�ɤ�����1�˶�Ť��ޤ���
- Add�ܥ����å����ƿ��������Ѥ��ɲä��ޤ���
- �������ɲä������Ѥ���֥륯��å��������뤤�Ф����ޤ������λ��Ѥ饤�ɤ�����2�˶�Ť��ޤ���
- ���ֱ�ü�����Ѥ���֥륯��å�����Ʊ�����뤤�Ф����ޤ���
- ����ˤ�ꡢ��-��-�Фο��γ�Ĵ�������Τ褦��ɽ������ޤ���
- ��ĴĴ���������������Ĥ��ơ����������Υ�������ǥͥåȥ����ΥΡ��ɤ����դ����줿���Ȥ��ǧ���Ƥ���������

�ǥե���ȤΥΡ��ɿ��Υԥ����Υ��ڥ��ȥ뤫��ȴ������Ƥ��뤳�Ȥ����դ��Ƥ����������褯�Ȥ�����ʤȤ���ȯ�����������ʤ��ͤϥ��ڥ��ȥ뤫����̤��������Ѥ��뤳�Ȥ�����ޤ���
- VizMapper�ѥͥ��Defaults�ͥåȥ�����������å����ޤ���
- Node Color��������NODE_FILL_COLOR�˥���ȥ�ǰŤ����������ޤ���
- �ͥåȥ���ӥ塼�ˤƥ����ॢ���Ȥ��Ƥ����Ĥ��ΥΡ��ɤ������ˤʤäƤ��뤳�Ȥ��ǧ���Ƥ���������
�����������ԤδĶ��ǤϥΡ��ɤη������꤬ȿ�Ǥ��줺��������
CREB kb�ȥ���ȥ������˦�֤���ӤǤ�����Ψ�Ѳ��ͤ�p�ͤ�ξ�����ɤ߹���Ǥ��ޤ���������p�ͤ�Ρ��ɤη��Ǽ����ޤ��礦�����������⤤��Τ�ͳѡ��㤤��Τ�ߤȤ��ޤ���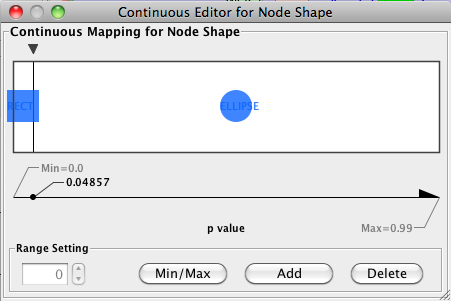
CREB kb�ȥ���ȥ������˦�֤���ӤǤ�����Ψ�Ѳ��ͤ�p�ͤ�ξ�����ɤ߹���Ǥ��ޤ���������p�ͤ�Ρ��ɤη��Ǽ����ޤ��礦�����������⤤��Τ�ͳѡ��㤤��Τ�ߤȤ��ޤ���
- Visual Mapping Browser��Unused Visual Properties��������Unused Properties�˥�����������Node Shape����֥륯��å����ޤ���
- ����ˤ��Node Shape��Visual Mapping Browser�Υȥåפ˰�ư���ޤ���
- Node Shape����������"Please select a value!"��å����ޤ���
- ����ˤ�����Ѳ�ǽ��°��̾���ɥ��åץ������˥塼��ɽ������ޤ���"p value"�����ޤ���
- Node Shape����������"Please select a mapping"��������"Please select mapping type"�ˤ�å����ޤ���
- ����ˤ�����Ѳ�ǽ�ʥ����פ��ɥ��åץ������˥塼��ɽ������ޤ���"Continuous Mapping"�����ޤ���
- ����ˤ��Node Shape����������"Graphical View"�˶��Υ�������������ޤ������Υ��������å����ޤ���
- continuous shape selection��������Continuous Editor for Node Shape�˥����������������ޤ���

- Add�ܥ����å����ޤ���
- ����ˤ�ꥹ�饤�������ΰ��Ⱦʬ��ʬ�������饤������ξ¦�˥Ρ��ɤη��Υ�������ɽ������ޤ���
- ��¦�ΥΡ��ɥ�������ʱ߷��ˤ���֥륯��å����ޤ���
- ����ˤ��Ρ��ɤη�������ݥåץ��åפ������ޤ���
- Rectangle������Apply�ܥ����å����ޤ���
- continuous shape selection��������Continuous Editor for Node Shape�˥����������˻ͳѤȱ߷��ΥΡ��ɤη��Υ�������ɽ������Ƥ��ޤ���
- �������ѥ���å����ƺ��˰�ư������0.5����侮�������ޤ�����������������ͤȤ��ޤ���
- continuous shape selection��������Continuous Editor for Node Shape�˥������������Ĥ��ơ������Ĥ��ΥΡ��ɤη����ͳѤ��̤ΥΡ��ɤ��߷��ˤʤä����Ȥ��ǧ���Ƥ���������
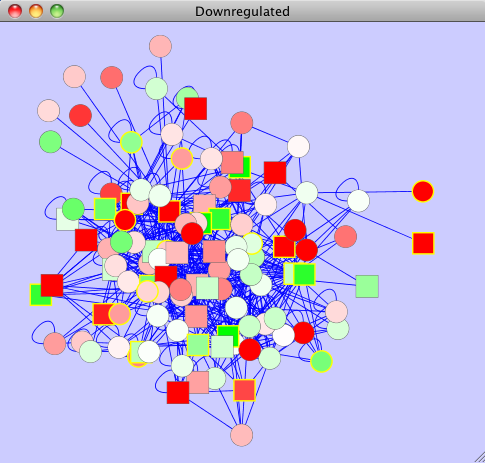
�����������ԤδĶ��ǤϥΡ��ɤζ��������꤬ȿ�Ǥ��줺��������
�Ρ��ɤζ������ȥ��������Ȥ����Ȥ��Ǥ���CREB��ʸ���ˤˤ��ͭ�դ����ͤ�Ķ�����Ρ��ɤ��ɤ��������Ȥ��Ǥ��ޤ������Υǡ����ϴ���°���Ȥ��ƥǡ������å�������Ѳ�ǽ�ȤʤäƤ��ޤ���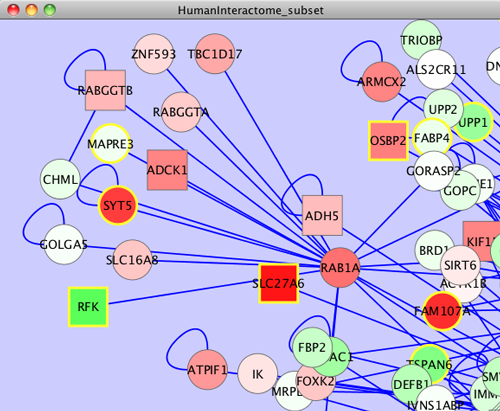
�Ρ��ɤζ������ȥ��������Ȥ����Ȥ��Ǥ���CREB��ʸ���ˤˤ��ͭ�դ����ͤ�Ķ�����Ρ��ɤ��ɤ��������Ȥ��Ǥ��ޤ������Υǡ����ϴ���°���Ȥ��ƥǡ������å�������Ѳ�ǽ�ȤʤäƤ��ޤ���
- Visual Mapping Browser��Unused Visual Properties����������Node Border Color����֥륯��å����ޤ���
- ����ˤ��Visual Mapping Browser�Υȥåפ�Node Border Color����ư���ޤ���
- Node Border Color����������"Please select a value!"��å����ޤ���
- ���Ѳ�ǽ��°��̾���ɥ��åץ������˥塼��ɽ������ޤ���"Sign CREB binding"�����ޤ���
- ���Ѳ�ǽ�ʥޥåԥ����פ��ɥ��åץ������˥塼��ɽ������ޤ���"Discrete Mapping"�����ޤ���
- �������Ԥ��Ǥ��ͤ�"yes"�ȤʤäƤ��ޤ�������°���ǤϤ����ͤΤߤ����Ѳ�ǽ�Ǥ���"yes"�α��ζ����å��������줿�ͳѤ����������������"..."��ɽ�����줿�ܥ���ˤ�å����ޤ���
- ��������̤�����ޤ������Υ���������������Ρ��ɤο��ˤ��Ф�����Ω�Ŀ������ޤ���������Ǥ����뤤���������ޤ����ͥåȥ����δ�Ϣ����Ρ��ɤ���������ɤ��ޤ���
- Visual Mapping Browser��Unused Visual Properties����������Node Line Width����֥륯��å����ޤ���
- ����ˤ��Visual Mapping Browser�Υȥåפ�Node Line Width����ư���ޤ���
- Node Line Width����������"Please select a value!"��å����ޤ���
- ���Ѳ�ǽ��°��̾���ɥ��åץ������˥塼��ɽ������ޤ���"Sign CREB binding"�����ޤ���
- ���Ѳ�ǽ�ʥޥåԥ����פ��ɥ��åץ������˥塼��ɽ������ޤ���"Discrete Mapping"�����ޤ���
- �������Ԥ��Ǥ��ͤ�"yes"�ȤʤäƤ��ޤ�������°���ǤϤ����ͤΤߤ����Ѳ�ǽ�Ǥ����ǥե���Ȥζ�����������1.5�ȤʤäƤ��ޤ���
- �������å�����5�����Ϥ��ޤ���
- �ͥåȥ���ΰ����˥����।��Ȱʲ��Τ褦�ˤʤäƤ���Ǥ��礦��

���Υ��������Ǥ�Cytoscape�ε�ǽ������Ĥ��Ҳ𤷤ޤ�������ϥͥåȥ���ȴ�Ϣ����ǡ�������Ϥ����ΤǤ���
�ǽ�ˡ������Ĥ����Ȥʤ�ǡ�����ɬ�פǤ����ҥȤι���������º�˦���μ¸��ǡ����Ǥ���cAMP���������祿��ѥ��Ǥ���CREB��shRNA�ˤ��Υå�������Υå��������˦��ȯ���ץ��ե������Ʊ����˦���Υ���ȥ������˦����Ӥ���Ƥ��ޤ���Pellegrini et al���ȡ�
�ǽ�ˡ������Ĥ����Ȥʤ�ǡ�����ɬ�פǤ����ҥȤι���������º�˦���μ¸��ǡ����Ǥ���cAMP���������祿��ѥ��Ǥ���CREB��shRNA�ˤ��Υå�������Υå��������˦��ȯ���ץ��ե������Ʊ����˦���Υ���ȥ������˦����Ӥ���Ƥ��ޤ���Pellegrini et al���ȡ�
�Ρ��ɤ����å���°���Τɤ��餫�˴�Ť���Cytoscape��Υͥåȥ���ϥե��륿���ݤ����뤳�Ȥ��Ǥ��ޤ��������ǡ�2���֤Ǥ���Ψ�Ѳ��ζ���˴�Ť����ͥåȥ���Υե��륿�ݤ���Ԥ��ޤ���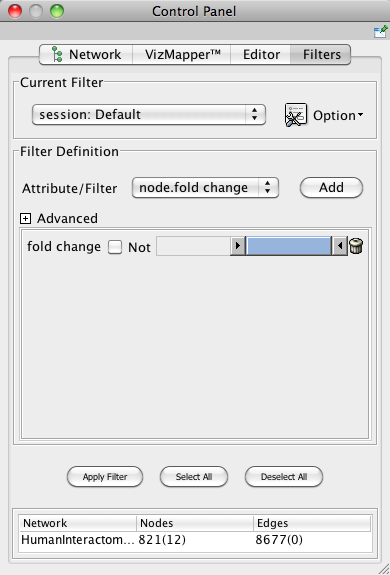
- Control Panel���Filters���֤�å����ޤ���
- Filter Definition���Attribute/Filter��"node.fold change"��å����ޤ���
- Filter Definition����������Add�ܥ����å���������°����ե��륿�����ɲä��ޤ���
- ����ˤ�ꡢ�ե��륿������Ψ�Ѳ��ˤĤ��ƤΥ��饤��������������ޤ���
- ���饤��������֥륯��å����ƥ��åȥ����ͤ����ޤ���low bound��2�����Ϥ���OK��å����ޤ���

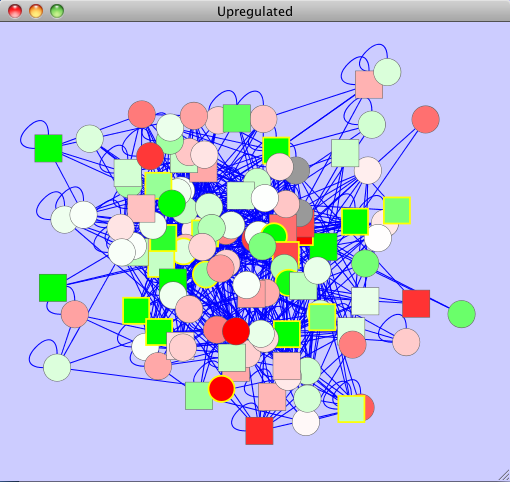
- �����ǡ������Ρ��ɤΤ����٤�ΥΡ��ɤ����뤿���Select �� Nodes �� First Neighbors of Selected Nodes�����ޤ���
- �������ͥåȥ����������뤿��ˡ�New �� Network �� From Selected Nodes, All Edges�����ޤ���
- force-directed�쥤�����Ȥ����ͥåȥ����Ŭ�Ѥ��뤿���Layout �� Cytoscape Layouts �� Force-Directed Layout�����ޤ���
- Control Panel��Network����ꤷ�ޤ����������ͥåȥ��̾���Խ����뤿��ˡ��ͥåȥ��̾����å�����Edit Network Title�����ޤ���"upregulated"�����Ϥ��ޤ���
- �������ͥåȥ���Ϥ��Τ褦�ˤʤ�Ǥ��礦��

- Repeat the Filter for down-regulated�����Ҥ˴ؤ��ƥե��륿���ݤ����֤�����Ψ�Ѳ���0.5̤���Ȥ��ޤ���2���ܤΥ��֥ͥåȥ����"downregulated"��̾�����դ��ޤ���
- "downregulated"�ͥåȥ���Ϥ��Τ褦�ˤʤ�Ǥ��礦��

�����ǡ��ͥåȥ������CREB1 (CREB)�Ρ��ɤ����Ƥߤޤ��礦��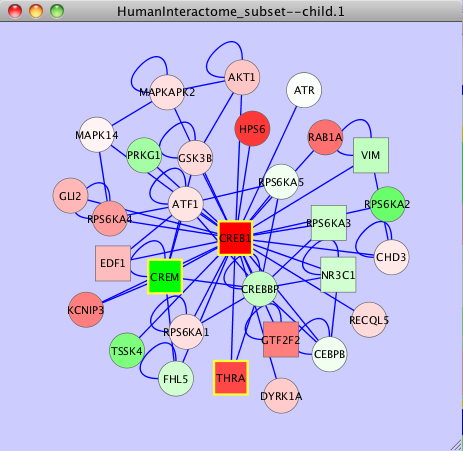
- �ġ���С��ˤơ�Search box�α��Ρ�Configure search options���������å����ޤ�.
- �����������������ˤơ��饸���ܥ����Nodes�������������ǥե���ȡˡ��ɥ��åץ������˥塼��Unique Identifier�����ޤ����������ǥե���ȡˡ�Apply�ܥ����å����ޤ���
- �����ե�����ɤˡ�"CREB"�����Ϥ��ޤ����ҥåȤ����ꥹ�Ȥ��������졢CREB1��̾�դ���줿�Ρ��ɤ����Ĥ���ޤ��������CREB transcription factor�Υ����ꥢ���Ǥ����ꥹ�Ȥ��餳�ΥΡ��ɤ������Enter���ޤ���
- �ͥåȥ�����CREB�Ρ��ɤ��ϥ��饤�Ȥ���ޤ���
- CREB�μ������ߺ��Ѥ�õ������Τ��ñ�ˤ��뤿��ˡ�CREB�ζ��٤Υͥåȥ�����˺������뤳�Ȥ��Ǥ��ޤ���Select �� Nodes �� First Neighbors of Selected Nodes�����ޤ���
- �Ρ��ɤΥ��åȤ��ϥ��饤�Ȥ���ޤ���File �� New �� Network �� From Selected Nodes, All Edges�����ޤ����������ͥåȥ������������ޤ���
- force-directed �쥤�����Ȥ�Ŭ�Ѥ��ƥͥåȥ�������ˤ��ޤ���
- �ͥåȥ���ϲ����Τ褦�ˤʤ�ޤ���

- �ͥåȥ���ڤ��뤳�Ȥˤ�ꡢCREB�����åץ쥮��졼�Ȥ�������쥮��졼�ȤΥΡ��ɤ���ߺ��Ѥ��Ƥ��뤫�뤳�Ȥ��Ǥ��ޤ���CREB��ž��������ž�̳�����ξ����Ư�����Τ��Ƥ��ޤ����⤷���åץ쥮��졼�Ȥȥ�����쥮��졼�ȤΥΡ��ɤ줾��ޤॵ�֥ͥåȥ����CREB������С�ξ���Υͥåȥ����CREB��¸�ߤ��뤳�Ȥ��狼��Ǥ��礦��
- ���ƥ��ꡧ
- ���䡦����
- �Х�������ե��ޥƥ�����

���Υڡ����ؤΥ�����
good
http://www.konosuke-matusita.com/
http://www.mbldaypassdeals.com/
http://www.erven-voor-ontwikkeling.com/
http://www.bossen-van-bezinning.com/
http://www.rustandbonescreenplay.com/
http://www.vivesolutionsinc.com/
http://www.darinkamontano.com/
http://www.awfmmellowtouch.net/
Best Canada Goose Jacket Canadian Goose Outlet Sale With Big Discount http://www.rustandbonescreenplay.com/
XxaGWZ Muchos Gracias for your blog article. Really Cool.
Cytoscape/Tutorials/Expression_Analysis_Human - �Х�������ե��ޥƥ��������� - Seesaa Wiki�ʥ������� for ���ޡ��ȥե��� モンクレール 2014 秋冬 レディース http://www.pslcbi.com/moncler2014.html
腕時計 ブランド [url=http://www.watchsrelation.com/]時計 ブランド メンズ[/url] <a href="http://www.watchsrelation.com/" title="時計 ブランド メンズ">時計 ブランド メンズ</a>
メンズ サングラス サングラス http://www.3stg.com/